Caractérisation biophysique des macromolécules et leurs complexes :
monodispersité, oligomérisation, masse moléculaire des protéines et leurs complexes, mesure thermodynamique des interactions.
Le plateau de Biophysique dispose de l’instrumentation et l’expertise pour la caractérisation des macromolécules et des interactions moléculaires en faisant appel à plusieurs méthodes biophysiques, incluant la diffusion de lumière statique et dynamique (DLS, SLS), le dichroïsme circulaire (CD), la fluorimétrie différentielle à balayage (DSF) et la titration calorimétrique isotherme (ITC). Ces méthodes permettent notamment de mesurer la solubilité, la stabilité, la masse molaire ainsi que l’état de polydispersité et d’oligomérisation des macromolécules et leurs complexes.
Ces équipements peuvent notamment être utilisés en amont d’une étude structurale ou d’un criblage pour caractériser les échantillons protéiques et vérifier qu’ils remplissent les conditions nécessaires à l’étude envisagée.
Elles sont également utilisées pour caractériser en détail le mode d’interaction de différents types de complexes macromoléculaires (interaction protéine-protéine (PPI), ligands, acides nucléiques) et suivre les changements conformationnels pouvant en résulter.
Prérequis
Toute étude biophysique requiert un échantillon protéique pur chimiquement. Cette pureté doit être validée à minima par analyse sur gel en conditions dénaturantes. Certaines méthodes, comme le microcalorimétrie demandent en plus une pureté oligomérique.
Offre de service
- DSF
- DLS & SEC-MALS
- ITC
- Dichroïsme Circulaire
Fluorimétrie différentielle à balayage (DSF)
C’est une méthode simple et rapide utilisée en format 96 (96 conditions testées simultanément) pour cribler différents tampons, pH, salinité, cofacteurs, acides nucléiques, …

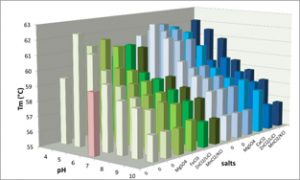
Son format haut débit est parfaitement adapté au criblage de petites chimiothèques de type fragments.
Dans cette méthode de criblage on mesure des changements dans la stabilité thermique de la protéine selon qu’elle soit seule ou en complexe avec un ligand.
DLS & SEC-MALS
La diffusion dynamique de la lumière (DLS) permet de caractériser l’homogénéité des solutions (mesure de la polydispersité), le taux d’agrégation et la taille des particules (protéines, ADN-ARN, complexes) par l’estimation du rayon hydrodynamique (Rh) des populations présentes.
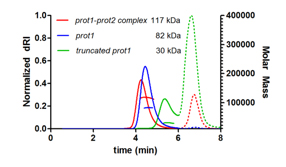
La chromatographie d’exclusion (SEC) couplée « en ligne » avec les mesures de la diffusion statique de la lumière à plusieurs angles (MALS), de l’index de réfraction (RI) et de l’absorbance (UV) est une méthode rapide et précise pour déterminer la masse molaire des protéines et des complexes en solution et l’état d’oligomérie. Elle permet également de contrôler l’homogénéité conformationnelle des échantillons.
L’ITC (titrage calorimétrique isotherme)
L’ITC ou (titrage calorimétrique isotherme) permet de mesurer la chaleur de réaction d’une solution en fonction du volume du réactif titrant ajouté, et de déterminer les paramètres thermodynamiques associés à l’interaction entre molécules de tout type (protéines, ADN, petites molécules…).
Le plateau de biophysique est équipé d’un calorimètre isotherme ITC200 entièrement dédié à l’étude des interactions moléculaires. Cet appareil permet de mesurer une constante d’association en solution et ceci sans nécessité de marquer les molécules. La gamme d’affinités nM-mM peut être mesurée de manière directe et des affinités de l’ordre du picomolaire peuvent être estimées grâce à des expériences de compétition.
Dichroïsme Circulaire (CD)
Dans le cas des protéines et des peptides, le dichroïsme circulaire permet d’évaluer le niveau de structuration de la macromolécule (repliement) et d’estimer les pourcentages de structures secondaires (hélices alpha, feuillet beta, random coil). La technique n’étant pas destructive il est possible d’effectuer des cinétiques ou titrations pour suivre les changements conformationnels. Il est également possible de déterminer la température de fusion (Tm) de la macromolécule et ceci sans la présence d’un fluorophore, ce qui rend la méthode CD complémentaire à la DSF. D’autres types de molécules sont analysables comme les acides nucléiques (ADN, ARN), les sucres et également les petites molécules.
Prérequis :
Macromolécule pure à ≥ 90%. La quantité et concentration dépendent essentiellement de la cuvette utilisée : de quelques µg/ml pour la cuvette 500µL (chemin optique 1mm) à quelques mg/ml pour la cuvette sandwich (chemin optique 0.01mm). Les meilleurs tampons sont le phosphate, le borate et le Tris-HCl. Eviter des fortes concentrations en NaCl et toutes molécules qui absorbent fortement dans l’UV.
L’ensemble de ces méthodes entre dans le cadre des prestations pour caractériser les macromolécules et leurs complexes. Selon vos besoins ou la nature du sujet, l’offre de service est large : mise à disposition des équipements (après une formation préalable), contrat de prestation de service ou collaboration scientifique.
- La DSF (fluorimétrie différentielle à balayage) est utilisée pour la détermination de la température de dénaturation d’une protéine. Cette méthode permet de cribler très rapidement différents conditionnements de protéine (pH, tampon, sel, …), ou d’étudier la formation de complexes (protéine/ligand, protéine/acide nucléique).
- La DLS et le SEC-MALS permettent de mesurer l’état d’oligomérisation et de déterminer la masse molaire de l’échantillon protéique.
- L’ITC permet de déterminer l’affinité et les bases thermodynamiques des interactions moléculaires.
- Le dichroïsme circulaire (CD) permet d’évaluer le repliement protéique et les structures secondaires d’un échantillon protéique.

