Identification de ligands innovants
Criblage de ligands par spectroscopie RMN, fluorimétrie différentielle à balayage (DSF) et cristallographie aux rayons X.
La plateforme PICT dispose de plusieurs chimiothèques (« drug-like », peptide, fragments) et offre l’accès et l’expertise à différentes méthodes complémentaires de criblage de ligands.
Ces méthodes reposent sur l’utilisation d’équipements et de méthodes de biophysique de pointe, telles que la RMN, la cristallographie aux rayons X et la fluorimétrie différentielle à balayage.
PICT vous propose ainsi de cribler ses chimiothèques ou vos propres banques de molécules. Notre expertise en RMN pourra être utilisée en amont ou en aval du criblage pour valider et/ou contrôler vos chimiothèques et déterminer la solubilité des composés.
- Criblage par RMN
- Criblage par DSF
- Criblage par cristallographie aux rayons X
Criblage par RMN
La spectroscopie RMN permet d’identifier des interaction protéine-ligand et donc s’inscrit comme une technique de choix dans le cadre d’une campagne de criblage pour l’identification de nouveaux ligands. La campagne de criblage s’opère selon deux étapes successives :
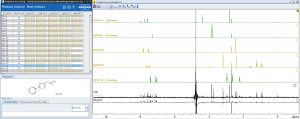
- Campagne initiale de criblage pour l’identification des ligands (touches) : différentes approches RMN très robustes (telles que STD, WaterLOGSY …) sont mises en œuvre pour identifier l’interaction de ligand d’affinité (µM à mM) avec une cible protéique, dans la cadre d’une approche par fragment.
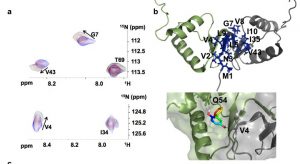
- Identification du site d’interaction : le site d’interaction peut être validé par une expérience de compétition (exp. STD, WaterLOGSY) avec un ligand connu ou en l’absence de compétiteur par une expérience de cartographie des déplacements chimiques de la protéine en présence du ligand. Outre l’identification du site d’interaction, cette dernière approche permet la construction d’un modèle d’interaction.
Criblage par DSF
La fluorimétrie différentielle à balayage (DSF) est une méthode robuste afin d’identifier des molécules susceptibles d’interagir avec une protéine, soit pour l’identification de partenaires physiologiques soit pour l’identification de touches dans la cadre d’un criblage de chimiothèques.
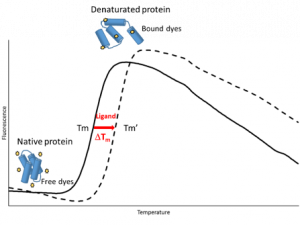
Les protéines sont incubées avec une sonde dont la fluorescence augmente lors d’interactions avec les régions hydrophobes des protéines. Le dépliement de la protéine, et l’exposition des parties hydrophobes, donnent alors naissance à un motif caractéristique de la fluorescence en fonction de la température.
Les applications concernent non seulement le criblage de ligands mais également l’identification de conditions optimales pour la purification et le stockage des protéines et de leurs partenaires pour faciliter la cristallisation. Du fait du peu de matériel protéique utilisé, du débit relativement élevé de la méthode et de la large gamme d’affinité couverte, la fluorimétrie différentielle à balayage est un outil précieux et parfaitement adapté pour le criblage par fragment.
Criblage par cristallographie aux rayons X
Lorsque les conditions de cristallisation d’une macromolécule sont connues, la détermination de structures par cristallographie aux rayons X peut être utilisée pour cribler des chimiothèques soit par co-cristallisation, soit par trempage.
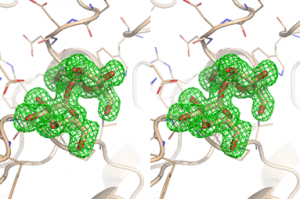
Les cristaux sont alors irradiés par rayonnement synchrotron et les données de diffraction collectées. Les densités électroniques obtenues dans la structure des complexe macromolécule/ligand sont analysées afin d’identifier les complexes potentiels macromolécule/ligand.
Les molécules interagissant de façon stable avec la macromolécule peuvent alors être identifiées. Cette technique présente l’avantage d’identifier des molécules interagissant de façon stable avec la protéine mais également de préciser les détails des interactions au niveau atomique.
Offre de service
Notre offre de service en biologie structurale couvre l’ensemble de nos expertises. Elle peut prendre differentes formes allant de la prestation de service à la collaboration scientifique. Une étude de faisabilité est souvent nécessaire.
La DSF (fluorimétrie différentielle à balayage) est une méthode très rapide permettant de cribler jusqu’à 1000 composés en 2 jours sur la base d’une interaction directe entre la cible et le ligand.
La spectroscopie RMN permet de cribler rapidement (en moins d’une semaine), une librairie de taille équivalente. Il est en plus possible de valider le site de fixation du ligand en utilisant par exemple un compétiteur déjà connu.
Lorsque les conditions de cristallisation de la cible sont connues, la cristallographie aux rayons X peut être utilisée comme méthode de criblage. Elle est moins rapide que la RMN mais présente l’avantage de fournir en même temps une identification du site d’interaction et une compréhension du mode d’interaction.
Une étude ambitieuse de découverte de ligands fait généralement appel aux trois approches selon une stratégie en trois étapes successives: criblage initial (ex. DSF et RMN), confirmation des touches (RMN) et caractérisation structurale du complexe ligand-protéine (cristallographie aux rayons X ou RMN).